да, кстати. коллеги, подскажите какой-нибудь простой набор операций (допустим, на пяток строк)
на котором можно было бы продемонстрировать профилирование?
а то обычно используют для этого графики ggplot (агрегация + визуализация), но я что-то не хочу их брать %(
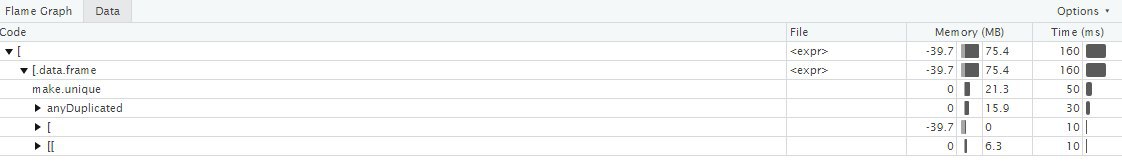
Как-то так у меня выглядело:
library(microbenchmark) # библиотека для сравнения скорости работы
# делаем довольно много данных
data(iris)
long_iris = iris[rep(seq_len(nrow(iris)), 1000), ]
# генерируем набор случайных индексов для сабсета
set.seed(123)
indices = sample(nrow(long_iris), 10000, replace = TRUE)
# цикл нужен, чтобы не слишком быстро считало - было время запрофилировать
# этой строчке делаем Profile selected lines
for(i in 1:100) long_iris[indices, ]
# без проверки на уникальность row.names
fast_subset = function(df, index){
res = lapply(df, "[", index)
class(res) = "data.frame"
rownames(res) = seq_along(res[[1]]) # нужна, иначе кол-во строк не будет определятся
res
}
# проверям, что одинаковый результат
temp1 = long_iris[indices, ]
temp2 = fast_subset(long_iris, indices)
all(temp1 == temp2)
# тестируем производительность
microbenchmark(
subset = long_iris[indices, ],
fast = fast_subset(long_iris, indices)
)
# выигрываем ~40% производительности
# Unit: microseconds
# expr min lq mean median uq max neval
# subset 1000.499 1043.3720 1269.918 1076.4790 1191.8575 15373.31 100
# fast 615.131 653.0385 1007.292 687.1395 729.8475 14622.77 100